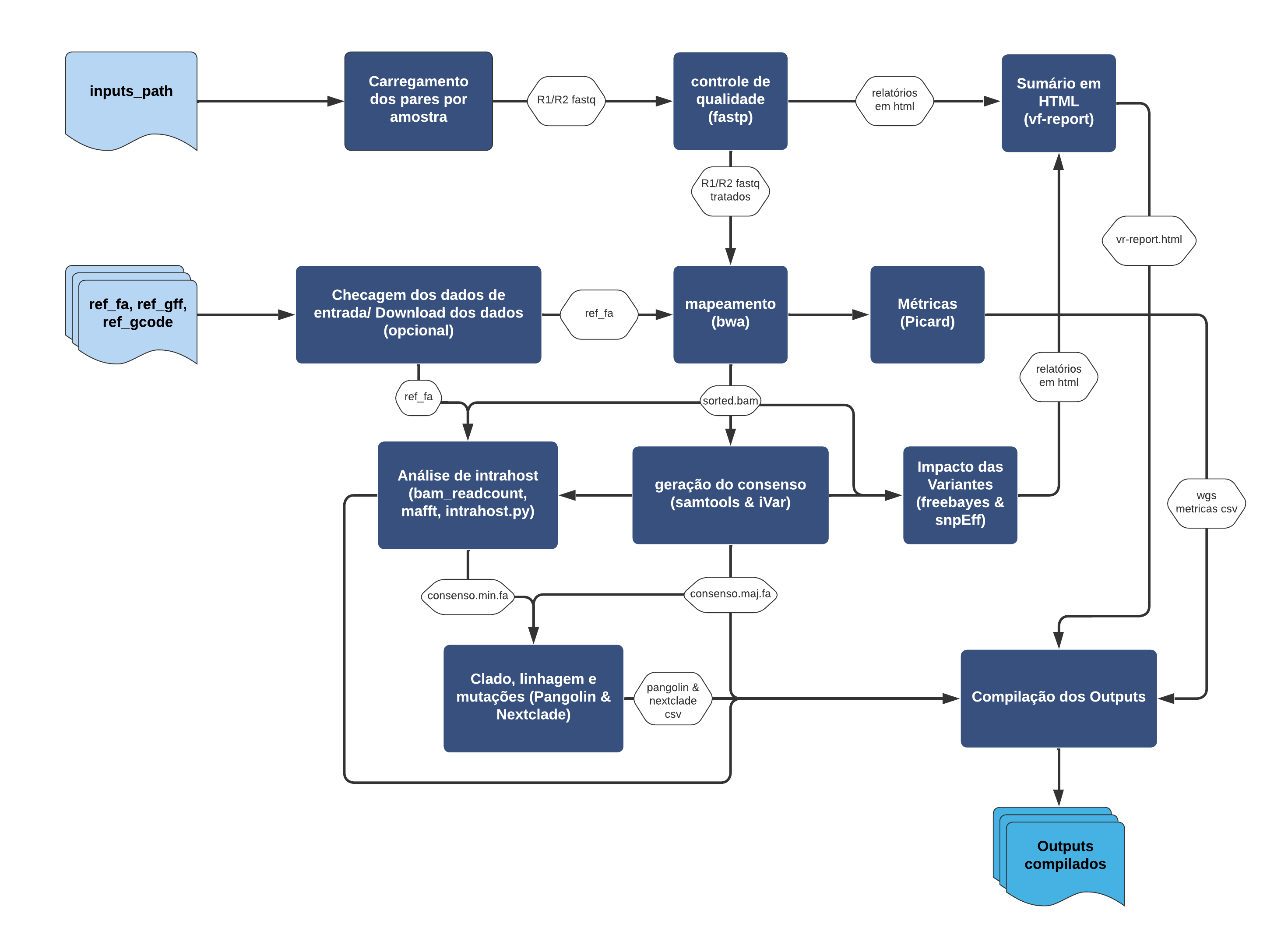
Sobre
ViralFlow é um workflow feito para profissionais da saúde, cujo objetivo é realizar todas as etapas de uma análise genômica viral por referência.
O código foi escrito na linguagem de workflow Nextflow, e pode ser aplicado para diferentes vírus, onde o usuário, após instalar a ferramenta, precisa rodar apenas 1 linha de código.
A equipe de desenvolvedores do ViralFlow não oferece suporte, até o presente momento, para erros relacionados à aplicação do código para outras plataformas. Atualmente, o código foi testado apenas para dados gerados por sequenciadores Illumina, utilizando estratégia de reads pareados (paired-end) ou não pareados (single-end).
A publicação da primeira versão do ViralFlow pode ser conferida neste link. A publicação da versão 1.0, que engloba as funções descritas nesta documentação, pode ser conferida neste neste link.
Análise Intrahospedeiro
O ViralFlow apresenta um algoritmo próprio para detectar regiões de iSNV (intrahost Single Nucleotide Variant).
Para quem um determinado loci multi alélico tenha um alelo em menor frequência considerado como iSNV, três condições precisam ser satisfeitas, estas condições estão esquematizadas na parte superior da figura abaixo.
Nesta lógica, a parte inferior da figura abaixo representa 3 sítios multi alélicos e exemplifica a lógica para considerar quais deles seriam considerados como iSNVs.

dependências
Para instalação e execução do ViralFlow, você precisará de pelo menos 4,4Gb de armazenamento de disco, para a criação dos contêineres, e de três dependências:
- pip
- Git
- uidmap
A lista com as versões de cada ferramenta utilizadas no ViralFlow pode ser conferida neste link. Além disso,para uma instalação com um ambiente controlado, utilizamos o Conda. O ViralFlow utiliza a ferramenta de containerizacao Singularity e da linguagem de workflow Nextflow. Para garantir o funcionamento correto do ViralFlow, é essencial as versões corretas do Singularity e do Nextflow, ambos podem ser instalados juntamente com o ViralFlow.
-
01 Conda
Conda é um gerenciador de pacotes de código aberto. Foi desenvolvido inicialmente para gerenciar ambientes de pacotes da linguagem Python, permitindo o controle de versões.
Para que todos os usuários utilizem o ViralFlow com as versões exatas do Singularity e do Nextflow, nós criamos um ambiente com estas versões, o que permite uma padronização nos ambientes de instalação do ViralFlow, bem como a resolução de eventuais erros.
-
02 Singularity v3.11.4
Singularity é uma ferramenta que executa a virtualização no nível do sistema operacional, tarefa popularmente conhecida como conteinerização.
Para uma pipeline computacional, como o ViralFlow, funcionar, ela precisa de várias dependências (outras ferramentas e bibliotecas do sistema operacional) e para evitar que o usuário precise instalar todas as dependências manualmente em sua máquina, nós usamos o sistema de containers.
Com a conteinerização, o usuário só precisa instalar uma ferramenta, neste caso o Singularity, e depois construir os containers com base em arquivos de receita (no caso do ViralFlow, isto também foi automatizado com o script setupContainers.sh). Dessa forma, diferentes grupos de pesquisa conseguem rodar a ferramenta com o mesmo ambiente computacional, sem mudanças no comportamento do ViralFlow, o que garante a reprodutibilidade dos resultados. -
03 Nextflow 22.04
Nextflow é um gerenciador de workflows de bioinformática que permite o desenvolvimento de workflows portáteis e reprodutíveis.
Nextflow permite a execução dos workflows em diversos ambientes computacionais, como ambientes locais, de computação de alta performance (HPC), serviços de computação na nuvem, como AWS e Google Cloud, e também em sistemas de orquestração de containers, como Kubernetes.
Além disso, o Nextflow provê suporte para gerenciamento de dependências com Conda, Docker, Podman ou Singularity.
Como Instalar (MacOS)?
Devido a limitações do uso do singularity no MacOS, para rodar o ViralFlow neste tipo de sistema, sugerimos a utilização de um software de virtualização Linux chamado Lima. Dessa forma, o usuário deve seguir três passos para instalação do Lima, e depois prosseguir com o guia de instalação do Ubuntu.
Instalando o Lima
Instale o Lima utilizando o gerenciador de pacotes Homebrew:
brew install lima
Instalando a instância Ubuntu
Siga o passo a passo da instalação de uma instância Ubuntu inserindo o comando abaixo
limactl start
Inicie o Ubuntu
Quando a máquina virtual for instalada, utilize o comando abaixo para iniciar o Ubuntu e siga os passos de instalação do ViralFlow normalmente.
lima
Como Instalar (Ubuntu)?
Para realizar a instalação do ViralFlow, são necessários quatro passos: Instalar as dependencies de sistema, caso você não as tenha instalado; Instalar o Conda; instalar o ViralFlow e por fim, montar os containers para as análises. Este processo é realizado uma única vez.
A instalação e uso do ViralFlow já foi testada nos sistemas:
- Ubuntu 20.04 LTS;
- Ubuntu 22.04 LTS;
Instalando as dependências do sistema
Caso você não tenha o instalador de dependencias pip, o sistema de controle de versões git, e o pacote uidmap, você deve realizar a instalação com as seguintes linhas:
sudo apt update -y && \
sudo apt upgrade -y && \
sudo apt install curl git python3-pip uidmap -y
Instalando e configurando o ambiente micromamba
Recomendamos o gerenciados de ambientes micromamba devido a sua paralelização ao fazer o download e instalação das dependências. Caso você utilize outro gerenciador de ambientes, como conda, miniconda, mamba, etc, você pode seguir a instalação do ViralFlow pulando esta etapa, porém, conflitos durante a instalação podem ocorrer devido as versões das dependencias especificadas, e suas respectivas disponibilidades em diferentes canais de recursos.
Caso você não tenha o gerenciador de ambientes micromamba instalado, você pode instalar e configurar com as seguintes linhas:
cd $HOME
curl -Ls https://micro.mamba.pm/api/micromamba/linux-64/1.5.7 | tar -xvj bin/micromamba
./bin/micromamba shell init -s bash -p ~/micromamba
source ~/.bashrc
micromamba activate
Instalando o ViralFlow
Caso você ja tenha as dependencias citadas e o conda instalado, você pode instalar o ViralFlow com 5 linhas de código:
git clone https://github.com/WallauBioinfo/ViralFlow
cd ViralFlow/
micromamba env create -f envs/env.yml
micromamba activate viralflow
pip install -e .
Construindo os containers
Todas as etapas do ViralFlow são executadas em ambientes controlados, onde cada ferramenta terá a mesma versão independente do grupo de pesquisa que esteja usando a ferramenta. O ViralFlow possui um método próprio para realizar toda essa construção de ambientes, rodando apenas uma linha de código. Para que a construção dos containers ocorra, o ViralFlow necessita que a ferramenta “unsquashfs” esteja disponível no diretório “/usr/local/bin/”. Durante o processo de construção dos containers, uma etapa de verificação da presença do unsquashfs é realizada. Caso o sistema do usuário não apresente o unsquashfs no local ideal, o ViralFlow irá sugerir os passos necessários para a solução deste problema. Para evitar este problema, o usuário pode criar um link sombólico da ferramenta “unsquashfs” para o diretório “/usr/local/bin/”, para isso o seguinte comando deve ser executado:
sudo ln -s /usr/bin/unsquashfs /usr/local/bin/unsquashfs
Após garantir que o unsquashfs esteja no local apropriado, rode o comando para a construção dos containers:
viralflow -build_containers
Como rodar (Guia Rápido)
O ViralFlow fornece 2 modos de uso: sars-cov2 e custom. Independente do modo, o usuário deve passar os caminhos absolutos (todo o caminho até o diretório ou arquivo a ser indicado para o pipeline ex. /home/usuario/teste/) para cada arquivo de entrada, caso contrário o pipeline será interrompido durante aexecução. A explicação de cada argumento pode ser conferida aqui.
Por padrão, a ferramenta snpEff presente no ViralFlow vem configurada apenas com o genoma NC_045512.2 do vírus SARS-CoV-2. Caso você queira incluir a análise do snpEff para outros vírus, você deve atualizar o banco do snpEff com a seguinte linha, exemplo com Dengue:
viralflow -add_entry_to_snpeff --org_name Dengue --genome_code NC_001474.2
sars-cov2
Neste modelo, a análise é realizada com base no genoma de referência NC_045512.2, e possui, como análises adicionais, a assinatura de linhagens com a ferramenta Pangolin, e a assinatura de clados e mutações com a ferramenta Nextclade. Para isto, o usuário precisa construir um arquivo com os parâmetros da análise, um exemplo pode ser conferido aqui.
viralflow -run --params_file test_files/sars-cov-2.params
custom
Neste modelo, a análise é realizada com base nos arquivos para o vírus que o usuário deseja analisar. Neste modo, o usuário é responsável por passar cada um dos arquivos necessários para a análise. Caso o usuário deseje realizar a análise do snpEff, deve passar o código do refseq do genoma viral para a análise:
viralflow -run --params_file test_files/denv.params
Atualização pangolin
Periodicamente a ferramenta pangolin atualiza o banco de linhagens, bem como a filogenia de classificação do usher, as constelações de mutações do scorpio, e o modelo treinado do pangoLearn. Para realizar a atualização da ferramenta e/ou de suas bases de dados, basta rodar o ViralFlow com algum dos comandos:
#atualiza a ferramenta e as bases de dados
viralflow -update_pangolin
#atualiza apenas as bases de dados
viralflow -update_pangolin_data
Argumentos
| Argumento | Valor Padrão (default) | Descrição |
|---|---|---|
| virus | sars-cov2 | Tipo de análise (sars-cov2 ou custom) |
| primersBED | null | Caminho absoluo do arquivo BED contendo informações dos primers usados na amplificação genomica (opcional). |
| outDir | launchDir/output/ | Caminho absoluto para o diretório onde serão armazeandos os resultados. |
| inDir | launchDir/input/ | Caminho absoluto para o dirretório com os dados de entrada (diretório com os arquivos FASTQ). |
| runSnpEff | true | Necessidade de rodar a ferramenta snpEff (true ou false). |
| writeMappedReads | true | Necessidade de gerar os arquivos FASTQ contendo as leituras de sequenciamento que mapearam no genoma de referência. |
| minLen | 75 | Tamanho mínimo que o read deve ter para nao ser eliminado pelo FastP. |
| depth | 5 | Profundidade mínima que uma região deve ser sequenciada para ser considerada no consenso. |
| mapping_quality | 30 | Limiar de qualidade de mapeamento para chamada de variantes. |
| base_quality | 30 | Limiar de qualidade de base para chamada de variantes. |
| minDpIntrahost | 100 | Profundidade mínima que uma região deve ter para ser considerada na análise de intrahost. |
| trimLen | 0 | Número de bases que devem ser cortadas (“trimadas”) em ambas as extremidades dos reads. |
| refGenomeCode | null | Código refseq do genoma a ser utilizado na análise custom. |
| referenceGFF | null | Arquivo GFF do genoma a ser utilizado na análise custom. |
| referenceGenome | null | Arquivo fasta do genoma a ser utilizado na análise custom. |
| nextflowSimCalls | null | Número de chamadas simultâneas que o nextflow pode realizar |
| fastp_threads | 1 | Número de threads a ser utilizado na análise da ferramenta fastp. |
| bwa_threads | 1 | Número de threads a ser utilizado na análise da ferramenta bwa. |
| dedup | false | Este argumento habilita o modo de desduplicação do fastp. para ativá-lo, altere o valor para true no arquivo de teste de parâmetros. |
| ndedup | 3 | Quando o modo de desduplicação está ativo, você pode usar níveis de precisão (1 - 6). Você pode alterar esse valor, mas recomendamos o padrão. Quanto mais alto, mais RAM e tempo são consumidos. para ativá-lo altere o valor de 1 para 6 no arquivo de teste de parâmetros. |
Arquivos de Outputs
A ferramenta ViralFlow produz 2 tipos de outputs: específico e compilado.
Específico
Para cada amostra na análise será criado um diretório de resultados com o padrão `prefixo_results` onde o prefixo é um código criado a partir do nome do arquivo fastq da amostra. Cada diretório conta com os seguintes resultados:
| Arquivo | Descrição |
|---|---|
| prefixo.all.fa.pango.out.csv | Arquivo tabular com os resultados da ferramenta pangolin. |
| prefixo.ann.vcf | Arquivo vcf em formato tsv (Tab separated value - valores separados por vírgula) com a anotação das variantes feita pela ferramenta snpEff. |
| prefixo.depth5.all.fa.nextclade.csv | Arquivo tabular com os resultados da ferramenta nextclade. |
| prefixo.depth5.amb.fa | Genoma consenso com nucleotídeos ambíguos em posições de alelos múltiplos. |
| prefixo.depth5.fa | Genoma consenso com nucleotídeos majoritários. Consenso de maioria, normalmente depositado no GISAID. |
| prefixo.depth5.fa.algn | Genoma consenso alinhado com o genoma de referência (seguindo o mesmo tamanho, mafft --keeplenght). |
| prefixo.depth5.fa.algn.minor.fa | Genoma consenso com nucleotídeos minoritários. |
| prefixo.depth5.fa.bc.intrahost.short.tsv | Arquivo tabular resumindo as posições genômicas onde as variantes intrahospedeiro aparecem. |
| prefixo.depth5.fa.bc.intrahost.tsv | Arquivo tabular com todas as informações das posições de variantes intrahospedeiro. |
| prefixo.mapped.R1.fq.gz | Arquivo FASTQ R1 com os reads mapeados. |
| prefixo.mapped.R2.fq.gz | Arquivo FASTQ R2 com os reads mapeados. |
| prefixo.metrics.genome.tsv | Arquivo tabular com métricas de profundidade e cobertura do mapeamento. |
| prefixo.fastp.html | Arquivo html com a sumarização dos resultados da ferramenta fastp. |
| prefixo_snpEff_summary.html | Arquivo html com a sumarização dos resultados da ferramenta snpEff. |
| prefixo.sorted.bam | Arquivo com o mapeamento sorteado dos reads da amostra contra o genoma de referência. |
| prefixo.tsv | Arquivo vcf-like gerado pelo iVar. |
| prefixo.unmapped.R1.bam.fq | Arquivo FASTQ R1 com os reads não mapeados. |
| prefixo.unmapped.R2.bam.fq | Arquivo FASTQ R2 com os reads não mapeados. |
| prefixo.vcf | Arquivo vcf. |
| metrics.alignment_summary_metrics | Arquivo de texto com um sumário de diversas métricas do mapeamento. |
| nextclade.errors.csv | Arquivo informando os erros do nextclade. |
| nextclade_gene_*.translation.fasta | Arquivo fasta com as proteínas de cada gene. |
| snpEff_genes.txt | Arquivo tabular com o número de variantes por impacto por gene. |
| wgs | Arquivo textual com métricas do mapeamento, incluindo profundidade por região. |
| prefixo_coveragePlot.png | Arquivo PNG com a visualização gráfica da cobertura do genoma. |
| prefixo_coveragePlot.svg | Arquivo SVG com a visualização gráfica da cobertura do genoma. |
| prefixo_coveragePlot.html | Arquivo HTML com a visualização gráfica da cobertura do genoma. |
| prefixo_snpPlot.png | Arquivo PNG com a visualização gráfica dos SNPs detectados na amostra em relação ao genoma referência. |
| prefixo_snpPlot.svg | Arquivo SVG com a visualização gráfica dos SNPs detectados na amostra em relação ao genoma referência. |
Compilado
Ao final da análise, é gerado um diretório nomeado COMPILED_OUTPUT com os resultados compilados, onde:
| Arquivo | Descrição |
|---|---|
| errors_detected.csv | Amostras que não foi possível gerar consenso. |
| lineage_summary.csv | É gerado em análises do sars-cov-2, apresenta a contagem das linhagens identificadas na análise, incluindo as linhagens de genomas minoritários. |
| major_summary.csv | Arquivo tabular com um sumário da profundidade, cobertura e linhagem (em caso de análises do sars-cov-2) dos consensos majoritários. |
| minor_summary.csv | Arquivo tabular com um sumário da profundidade, cobertura e linhagem (em caso de análises do sars-cov-2) dos consensos minoritários. |
| mutations.csv | Arquivo tabular com as mutações encontradas. |
| nextclade.csv | Arquivo tabular com o resultado do nextclade (em caso de análises do sars-cov-2). |
| pango.csv | Arquivo tabular com o resultado do pangolin (em caso de análises do sars-cov-2). |
| reads_count.csv | Arquivo tabular com a contagem de reads por amostra. |
| seqbatch.fa | Arquivo fasta com os consensos majoritários. |
| short_summary.csv | Arquivo tabular com um sumário da profundidade, cobertura e linhagem (em caso de análises do sars-cov-2) de ambos os consensos, com as seguintes colunas:
|
| wgs.csv | Arquivo tabular com o resultado do picard. |
| VF_REPORT/index.html | Arquivo html com a compilação dos resultados do fastp e do snpEff. |
Perguntas Frequentes (FAQ)
Devo montar os containers a cada análise?
Não, os containers só devem ser reconstruídos caso ocorra alguma atualização na ferramenta.
O ViralFlow retornou um erro devido a falta do "unsquashfs" no diretório "/usr/local/bin/" o que devo fazer?
Um link simbólico para o "unsquashfs" deve ser criado, para isso rode a seguinte linha de comando: sudo ln -s /usr/bin/unsquashfs /usr/local/bin/unsquashfs
Eu devo fornecer o arquivo fasta de adaptadores?
Você deve fornecer o arquivo fasta com os primers de PCR apenas em análises que utilizaram PCR para amplificação do material genético viral antes do sequenciamento.
Minha análise do ViralFlow reportou um erro na etapa de atualização do Pangolin, por que?
Isto pode ocorrer caso a equipe do Pangolin lance uma versão com novas dependências, neste caso, você deve remontar o container do pangolin com a linha: singularity pull docker://staphb/pangolin
Minha instalação deu errado na etapa de montar os containers, por que?
O que pode ter acontecido é que você tenha uma versão do singularity diferente da versão v3.11.4, você pode checar isso com singularity --version. Outro fator que pode ocasionar erros é a sua rede de internet bloquear os dominíos de consulta para construção dos containers.
Instalei a ferramenta, meus arquivos estão todos organizados seguindo o manual, mas as amostras não são encontradas, por que?
O ViralFlow reconhece amostras pelos sufixos _R1 ou _R2 seguido de .fq.gz ou .fastq.gz, certifique-se que suas amostras seguem o padrão necessário. Exemplos:
- ART1_R1.fq.gz, ART1_R2.fq.gz será reconhecido como a amostra ART1;
- ART1_denv_R1.fastq.gz, ART1_denv_R2.fastq.gz será reconhecido como a amostra ART1_denv.
Posso rodar o ViralFlow no Windows??
Apesar de algumas versões do sistemas Windows integrarem uma interface linux pelo WSL (Windows Subsystem for Linux), nossa equpe de desenvolvimento não testou o ViralFlow nestes ambientes, dessa forma, o usuário é livre para testar o workflow neste tipo de ambiente, porém nossa equipe não dará suporte para erros provenientes desses testes.
Como citar?
Atualmente existem duas publicações do ViralFlow, a primeira, de 2022 que engloba as versões "pré" 1.0, e a segunda de 2024 que engloba a versão 1.0
Dezordi, F.Z.; Neto, A.M.d.S.; Campos, T.d.L.; Jeronimo, P.M.C.; Aksenen, C.F.; Almeida, S.P.; Wallau, G.L.; on behalf of the Fiocruz COVID-19 Genomic Surveillance Network. ViralFlow: A Versatile Automated Workflow for SARS-CoV-2 Genome Assembly, Lineage Assignment, Mutations and Intrahost Variant Detection. Viruses 2022, 14, 217. https://doi.org/10.3390/v14020217
Silva, A. F., Silva Neto, A. M., Aksenen, C. F., Jeronimo, P. M. C., Dezordi, F. Z., Almeida, S. P., Costa, H. M. P., Salvato, R. S., Campos, T. L., Wallau, G. L., on behalf of the Fiocruz Genomic Network. (2024). ViralFlow v1.0—a computational workflow for streamlining viral genomic surveillance. NAR Genomics and Bioinformatics, 6(2), lqae056. https://doi.org/10.1093/nargab/lqae056
